GECCO Datensatz
Hier erhalten Sie alle Informationen zum GECCO Datensatz.
GECCO auf einen Blick
Zur Bewältigung der aktuellen Pandemie und der damit einhergehenden Behandlung von Patienten fördert das Bundesministerium für Bildung und Forschung (BMBF) ein nationales Netzwerk der Universitätsmedizin im Kampf gegen COVID-19. Unter anderem soll das Netzwerk die Daten der behandelten COVID-19 Patienten systematisch erfassen und bündeln. Die Forschenden sollen die Behandlung der COVID-19-Patienten standardisiert erheben, verfolgen und analysieren.
Die hohe Bedrohungslage hat zu intensiver wissenschaftlicher Aktivität zu COVID-19 geführt, wozu zahlreiche regionale, nationale und internationale epidemiologische Erhebungen und Registerstudien zählen. Ohne eine koordinierte Aktion und die Sicherstellung einer semantischen und syntaktischen Interoperabilität, einer gemeinsamen Sprache der erhobenen Daten, droht gerade die Vielzahl der Bemühungen eine gefährliche Segmentierung der Informationen zu erzeugen. Diese könnte im Ergebnis dringend benötigte wissenschaftliche Erkenntnisse verzögern oder gar verhindern.
Der Konsensusdatensatz gibt der Wissenschaft um COVID-19 eine gemeinsame Sprache und Arbeitsgrundlage.
Der GECCO Datensatz wurde kreiert, um Wissenschaftlern eine gemeinsame Grundlage zu geben, um die Zusammenführung und Verarbeitung von studienbezogenen Daten für die weitere Nutzung in allen beteiligten Projekten zur Verfügung zu stellen. Schon heute ist der GECCO Datensatz ein wichtiges Tool für die Projekte NAPKON, CODEX, COMPASS und ORCHESTRA.
Seit Mai 2020 ist der GECCO Datensatz für das gesamte Netzwerk Universitätsmedizin (NUM) und die allgemeine Öffentlichkeit auf ART-DECOR und Simplifier zugänglich.
Inhaltliche Parameter
- Anamnese/Risikofaktoren: Erfasst wird beispielsweise, ob der Patient/die Patientin Vorerkrankungen wie Herz-Kreislauf- oder Tumorerkrankungen, HIV oder Diabetes hat. Außerdem wird nach Risikofaktoren wie Rauchen gefragt.
- Bildgebung: Erfasst werden Daten bildgebender Verfahren wie z.B. von Röntgenbildern der Lunge.
- Demographie: Es werden z.B. Angaben zum Geburtsdatum, Geschlecht, Gewicht, zur Größe sowie zur ethnischen Zugehörigkeit erhoben.
- Epidemiologische Faktoren: Gefragt wird, ob der Patient/die Patientin in den letzten 14 Tagen vor Beginn seiner/ihrer Beschwerden wissentlich Kontakt mit einer wahrscheinlich oder nachgewiesenermaßen an COVID-19 erkrankten Person hatte.
- Komplikationen: Erhoben wird beispielsweise, ob der Patient/die Patientin eine Lungenembolie, einen Herzinfarkt oder eine Blutstrominfektion hat/hatte.
- Krankheitsbeginn/Aufnahme: Erhoben wird der Zustand bei der Diagnose/Aufnahme des Patienten/der Patientin.
- Laborwerte: Verschiedene Laborwerte werden erfasst wie z.B. Blutwerte.
- Medikation: Gefragt wird nach eingenommenen Medikamenten sowohl zur COVID-Therapie als auch zur Behandlung weiterer Erkrankungen.
- Outcome bei Entlassung: Erfasst wird die Art der Entlassung einer Patientin/eines Patienten, ob sie/er beatmet wurde und ob sie/er mit einem negativen oder positiven Testergebnis entlassen wurde.
- Studieneinschluss/Einschlusskriterien: Als Kriterium für die Teilnahme an der Studie wird erfasst, ob COVID-19 Hauptursache für die Aufnahme ins Klinikum ist und ob eine unterschriebene Einwilligungserklärung vorliegt.
- Symptome: Unter anderem werden Symptome wie Geruchs- bzw. Geschmacksstörungen, Bauchschmerzen, Bewusstseinsstörungen/Verwirrtheit, Durchfall, Erbrechen, Husten, Kurzatmigkeit, Übelkeit, Fieber und Kopfschmerzen erfasst.
- Therapie: Angegeben wird, welche Therapie der Patient/die Patientin erhält/erhalten hat z.B. Dialyse, Apherese, ECMO-Therapie, Behandlung auf der Intensivstation.
- Vitalparameter: Erfasst werden Werte wie Körpertemperatur, Herz- und Atemfrequenz, Blutdruck und Sauerstoffsättigung
Unsere Geschichte
Basis des GECCO Datensatzes sind sowohl die Studie Pa-COVID-19, das ISARIC-Protokoll der WHO als auch die Datenkonzepte des LEOSS-Registers.
PA-COVID-19 ist eine zentrale Phänotypisierungs-Plattform und longitudinale Registerstudie für COVID-19-Patienten an der Charité in Berlin. Sie dient der harmonisierten klinischen und molekularen Phänotypisierung von COVID-19-Patient:innen.
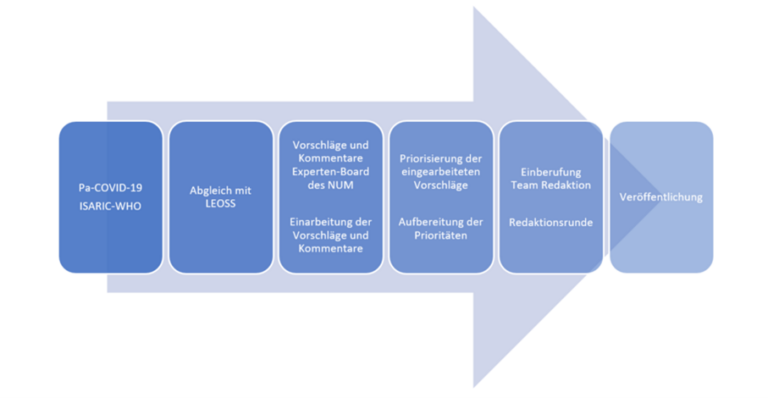
Der Zweck von ISARIC besteht darin, Krankheiten und Todesfälle durch Ausbrüche von Infektionskrankheiten wie COVID-19 zu verhindern. In ISARIC haben sich weltweit klinische Forschungsnetzwerke zusammengeschlossen, um eine möglichst rasche Forschungsreaktion beim Ausbruch einer Infektionskrankheit bieten zu können.
Die ESCMID Emerging Infections Task Force (EITaF), das Deutsche Zentrum für Infektionsforschung (DZIF) und die Deutsche Gesellschaft für Infektiologie haben das Netzwerk LEOSS initiiert, um ein klinisches Patientenregister für mit SARS-CoV-2 infizierte Patient:innen einzurichten. Das Ziel von LEOSS ist die flächendeckende und sektorenübergreifende Erfassung der klinischen Epidemiologie von Patienten mit COVID-19, mit dem Ziel die Erkrankung besser zu verstehen, prognostische Faktoren zu erkennen und gängige Interventionen zu bewerten, um die Patientenversorgung zu verbessern.
Aktueller Stand und Implementierung
Es wurden ca. 80 Datenkonzepte mit 280 Antwortmöglichkeiten nach festgelegten Kriterien in einem transparenten Prozess ausgewählt. Die semantische Annotation in SNOMED CT, LOINC, UCUM, ICD und ATC sowie die beginnenden Arbeiten zur FHIR-Modellierung sind online jederzeit abrufbar.
Antwortmöglichkeiten, Referenzbereiche, Maßeinheiten und semantische Annotationen wurden in der detaillierten Beschreibung in ART-DECOR® aufgeführt.
Die Entwicklung wurde in enger Zusammenarbeit mit den Fach- und Organspezifischen Arbeitsgruppen (FOSAs) und dem Fachbeirat abgestimmt, was das Monitoring und die Qualitätskontrolle des Prozesses gewährleistet und sicherstellt.
Die Übersicht über die einzelnen Parameter und Antwortmöglichkeiten finden Sie in unserer ausführlichen Beschreibung unter Publications.
GECCOplus
Im Rahmen von NAPKON bildet der GECCO-Kerndatensatz den Minimaldatensatz aller drei NAPKON-Kohorten HAP (hochauflösende Plattform), SÜP (sektorenübergreifende Plattform) und POP (populationsbasierte Plattform). Kohorten-übergreifend existieren ungefähr 2000 Datenelemente. Die Schnittmenge aller drei NAPKON-Kohorten gründen den GECCOplus-Datensatz. Die Identifikation der Schnittmengen erfolgte auf Basis der einzelnen Kohortendatensätze sowie eines programmierten Python-Skriptes unter Verwendung der Levenshtein-Distance.
Der GECCO83 ist wesentlicher Bestandteil aller drei Kohortendatensätze und somit bereits in jedem Kohortendatensatz enthalten. Um die Dopplung von Datenelementen im GECCOplus zu vermeiden, wurde der erzeugte Output um diese Datenelemente entsprechend bereinigt. Die daraus resultierende Schnittmenge zweier Kohorten (z.B. SÜP und POP) wurde der dritten Kohorte (z.B. HAP) gegenübergestellt.
Schlussendlich bildet der letzte Abgleich die Definition des GECCOplus-Datensatzes und somit die Schnittmenge aller drei Kohortendatensätze. Um den GECCOplus standardisiert abzubilden und aufzubereiten wurde eine einheitliche Bezeichnung der Items mit Hilfe von SNOMED-CT und Medical Subject Headings (MeSH) vorgenommen. Außerdem wurden die Antwortausprägungen durch die Redaktionsgruppe des GECCO83 festgelegt.
GECCO Module
Die Entwicklung des GECCOplus Datensatzes wird für das Projekt NAPKON in enger Zusammenarbeit und Abstimmung mit den Kohorten HAP (Hochauflösende Plattform), SÜP (Sektorenübergreifende Plattform) und POP (Populationsbasierte Plattform) erstellt. Dieser Datensatz soll eine Schnittmenge der bereits vorhandenen Datensätze der einzelnen Kohorten bilden. So können alle drei Kohorten auf einen gemeinsamen Datensatz neben den kohortenspezifischen Datenparametern zugreifen.
Der GECCO Datensatz wurde neben GECCOplus um drei fachspezifische Module ausgebaut. Dies geschah in sehr enger Zusammenarbeit und ständigem Austausch mit den FOSA und weiteren Spezialisten. Die Module beinhalten spezifischere Parameter, welche es ermöglichen detaillierte und tiefergreifende Daten fachspezifisch zu erheben.
In 2021 lag der Fokus auf der Entwicklung von drei Modulen:
- Pädiatrie Modul
- Kardiologie Modul
- Impfmodul
GECCO ist designed um reaktionsschnell einen harmonisierenden Datenaustausch mit Real World Data Plattformen, Gesundheitsämtern und internationalen Partnern zu ermöglichen.
Da GECCO auf internationalen Standards und Terminologien beruht, ist die semantische und syntaktische Interoperabilität in unterschiedlichen Systemen gewährleistet.
Publikationen
The German Corona Consensus Dataset (GECCO): a standardized dataset for COVID-19 research in university medicine and beyond.
Sass J., Bartschke A., Lehne M., Essenwanger A, Rinaldi E, Rudolph S, Heitmann KU, Vehreschild JJ, von Kalle C, Thun S
BMC Med Inform Decis Mak 20, 341 (2020). DOI
